COVID-19: ¿Cuándo es seguro dar de alta a pacientes que han sido infectados por el nuevo coronavirus?
Un nuevo estudio del hospital universitario más grande de Europa, La Charité – Universitätsmedizin de Berlín, en conjunto con la clínica Schwabing de Munich y el Instituto Bundeswehr de Microbiología en Alemania, logró establecer el lugar preferente de replicación del nuevo coronavirus y el momento en que los pacientes que han desarrollado la enfermedad ya no son contagiosos y pueden ser dados de alta en forma segura.
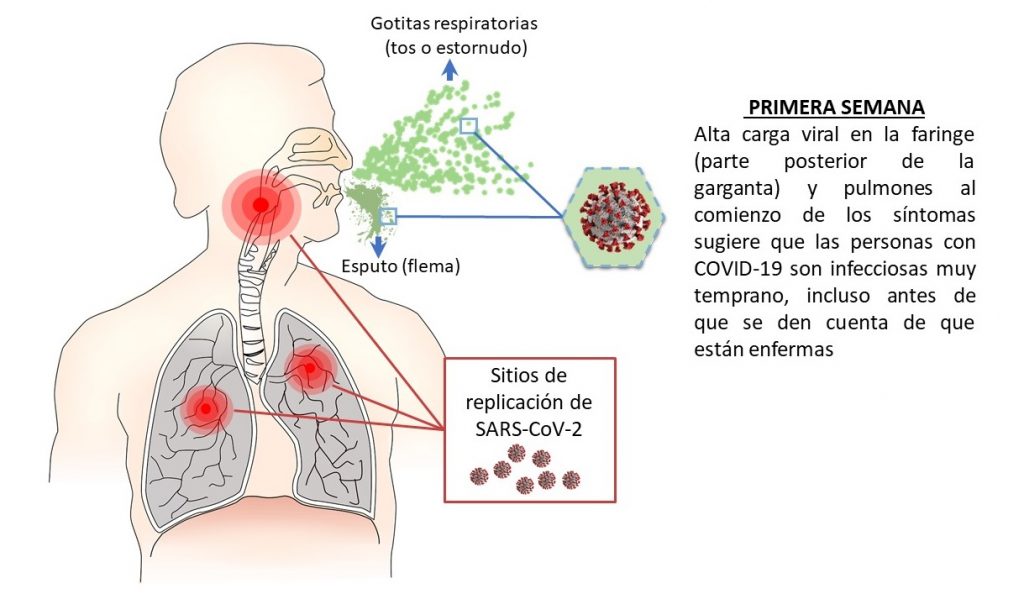
El primer brote de la enfermedad por el nuevo coronavirus (COVID-19) en Alemania se descubrió el 27 de enero de este año, en un grupo de pacientes del área de Starnberg, cerca de Munich. Nueve de estos pacientes recibieron tratamiento en la clínica Schwabing y fueron estudiados durante el curso de su enfermedad. El equipo de investigación publicó recientemente sus hallazgos en la prestigiosa revista Nature, donde comparten información clave para las estrategias de salud pública implementadas en los países que enfrentan la pandemia, como, por ejemplo, que el virus se replica principalmente en la faringe, esto significa que el nuevo coronavirus no tiene que viajar a los pulmones para replicarse, por lo que es muy fácil que las personas infectadas transmitir el virus por gotitas respiratorias, no solo antes de mostrar síntomas, sino también después de que los síntomas hayan desaparecido.
El estudio
Esta investigación proporciona el primer análisis detallado de la dinámica de la infección por el nuevo coronavirus SARS-CoV-2 en pacientes con enfermedad leve. Los pacientes analizados eran jóvenes o de mediana edad y presentaban síntomas leves parecidos a la gripe, como tos, fiebre, pérdida del gusto y el olfato, además no tenían enfermedades subyacentes significativas. Todos los casos adquirieron sus infecciones por contacto con un caso inicial, lo que significa que no se estudiaron simplemente en función de la presencia de síntomas. Durante la investigación, a todos los pacientes se les tomaron muestras desde la nariz y la faringe, con hisopos nasofaríngeos (parecidos a los «cotonitos» pero de uso clínico), y de esputo (flema) a partir del día 1, cuando se iniciaron los síntomas, hasta el día 28. Posteriormente, el equipo de investigación detectó el SARS-CoV-2 en todas las muestras recolectadas a través de la técnica de RT-PCR (para detalles del método, ver infografía aquí). Los análisis se realizaron en dos laboratorios separados que trabajan de forma independiente: el Instituto de Virología en el Campus Charité Mitte en Berlín y el Instituto Bundeswehr de Microbiología, institución que forma parte del Centro Alemán para la Investigación de Infecciones (DZIF, por sus iniciales en alemán). Para descartar la coinfección de los pacientes con otros virus respiratorios que pudieran explicar o agravar los síntomas, también analizaron la presencia de otros agentes infecciosos respiratorios en las muestras, incluyendo: cuatro coronavirus diferentes (HCoV-HKU1, -OC43, -NL63, -229E), virus de influenza A y B, rinovirus, enterovirus, virus sincicial respiratorio, virus de parainfluenza humana 1-4, metapneumovirus, adenovirus y bocavirus humanos. Ninguno de estos virus fue detectado en los nueve pacientes analizados. El equipo de investigación también tomó muestras diarias de orina y de suero, pero resultaron negativas para el ARN del SARS-CoV-2, mientras que en muestras de heces sí fue posible detectar el virus. El análisis de los hisopos tomados entre los días 1 y 5 de todos los pacientes resultaron positivos para SARS-CoV-2 con una alta carga viral (número de copias de ARN viral). Los que fueron tomados después del día 5 tenían alrededor del 50 % de la carga viral promedio observada en los hisopos de los primeros días y una tasa de detección de 39,93 %. La carga viral promedio en el esputo también fue alta y ligeramente superior a lo observado en los hisopos nasofaríngeos en los primeros 5 días (alrededor de un 3,5% mayor). Mientras que los síntomas disminuyeron principalmente hacia el final de la primera semana, el ARN viral permaneció detectable en los frotis de garganta hasta la segunda semana. Los dos pacientes que mostraron algunos signos de infección pulmonar fueron los únicos casos en que las cargas virales de esputo mostraron un pico alto y tardío alrededor del día 10/11, mientras que las cargas virales de esputo estaban disminuyendo en ese momento en todos los demás pacientes. Las muestras de heces y esputo permanecieron con ARN SARS-CoV-2 positivo durante tres semanas en seis de los nueve pacientes, a pesar de la resolución completa de los síntomas.
Infectividad
El virus requiere infectar una célula para replicarse, allí produce nuevas partículas virales, las que posteriormente salen de la célula e infectan nuevas células mediante la unión a receptores celulares específicos. El equipo de investigación utilizó células Vero E6 ―células epiteliales de riñón extraídas de un mono verde africano―, para aislar el virus en forma controlada en los laboratorios. Luego de infectar las células con las muestras clínicas obtenidas de los pacientes, se tomaron muestras de los cultivos celulares después de 0, 1, 3 y 5 días y se utilizaron en análisis de RT-PCR. De una fracción considerable de muestras (16.66% en hisopos, 83.33% en muestras de esputo) el virus se aisló fácilmente durante la primera semana de síntomas y no se obtuvieron aislamientos de las muestras tomadas después del día 8, a pesar de la alta carga viral. El aislamiento exitoso desde los hisopos de garganta tempranos sugirió al equipo de investigación que la replicación del virus ocurría en los tejidos del tracto respiratorio superior, es decir, en la faringe. Para obtener pruebas de la replicación activa del virus realizaron análisis de RT-PCR para identificar los ARN mensajeros subgenómicos virales (sgARN) directamente en las muestras clínicas. El sgRNA viral solo se transcribe en células infectadas y no se empaqueta en viriones (las partículas virales que salen de las células para infectar a otras), lo que indica la presencia de células infectadas activamente en las muestras. Los resultados demuestran la replicación activa de SARS-CoV-2 en la garganta durante los primeros cinco días después del inicio de los síntomas. También se hicieron análisis en las heces de los pacientes, pero o no se encontró sgRNA viral en la mayoría de las muestras, o en algunas los niveles fueron muy mínimos. Estos datos confirman que la forma infecciosa del virus se aísla fácilmente de muestras derivadas de garganta y pulmón, pero no de las heces. Estos resultados sugieren que las medidas para contener la propagación viral deberían apuntar a la transmisión basada en gotitas, persona a persona, en lugar de a vectores pasivos como las superficies, donde según el estudio existe un menor riesgo de infecciosidad. El desprendimiento prolongado del virus en el esputo sugiere que los pacientes deben permanecer aislados en casa después del alta hospitalaria.
Presencia de anticuerpos
Mediante inmunofluorescencia se analizó la presencia de las inmunoglobulinas G y M, a través de una prueba de neutralización (ver infografía) en células que expresaban la proteína espiga del SARS-CoV-2, proteína de la superficie viral que es responsable de la entrada del virus en la célula huésped. Utilizando muestras de sangre de los pacientes con COVID-19, esta prueba detecta los anticuerpos que neutralizan el virus SARS-CoV-2, es decir, los que detienen el virus que infecta las células. Los resultados muestran que la mitad de los pacientes evaluados habían desarrollado anticuerpos al día 7 después del inicio de los síntomas. Después de dos semanas se detectaron anticuerpos en todos los pacientes. El inicio de la producción de anticuerpos coincidió con una disminución gradual de la carga viral, esto ofrece la esperanza de que una vacuna, dirigida a la proteína espiga del virus, pueda ser efectiva para prevenir la infección.
Referencia
Wölfel, R., Corman, V.M., Guggemos, W. et al. Virological assessment of hospitalized patients with COVID-2019. Nature (2020). https://doi.org/10.1038/s41586-020-2196-x
